Une nouvelle technique pour accélérer la génétique

L' appareil SMiLE-Seq © A. Isakova/EPFL
Des scientifiques de l'EPFL ont développé une technique susceptible de changer la donne en génétique, en rendant la caractérisation des protéines de liaison d'ADN plus rapide, plus précise et efficace.
Les gènes détiennent le code génétique pour produire l'ensemble des protéines de la cellule. Pour commencer ce processus, les gènes doivent être transcrits d'ADN en ARN. Cela exige une vaste famille de protéines de liaison nommées facteurs de transcription. En raison de leur importance dans l'expression des gènes, ceux-ci présentent un énorme intérêt pour les biologistes. Toutefois, en raison même de leur nombre, de leur capacité à se combiner en des paires différentes et la difficulté technique que présente l'étude de leurs propriétés de liaison à l'ADN en laboratoire, les facteurs de transcription sont méconnus. Des scientifiques de l'EPFL ont développé une technique basée sur la microfluidique susceptible d'accélérer fortement le processus avec peu de matériel. Ils ont déjà eu recours à celle-ci pour déterminer les propriétés de liaison d'ADN de plus de 60 facteurs de transcription, avec parmi eux neuf nouveaux facteurs. Leurs travaux sont publiés dans Nature Methods.
Facteurs de transcription
Les mammifères – humains compris – ont entre 1300 et 2000 facteurs de transcription, dont beaucoup se combinent avec d'autres en «hétérodimères», de manière à lier les gènes et induire leur transcription en ARN. Les hétérodimères sont estimés entre 3'000 et 25'000.
En conséquence, le nombre de combinaisons peut être très élevé. Comprendre leurs propriétés de liaison d'ADN, ainsi que leur affinité et leur spécificité pour l'ADN est un aspect-clé pour exploiter à l’avenir les facteurs de transcription à des fins biotechnologiques ou pharmaceutiques.
Plusieurs bases de données lisent les propriétés de liaison d'ADN, mais elles ne couvrent que quelque 500 facteurs de transcription uniques, et seulement une fraction d'hétérodimères. Dans ce domaine, les progrès sont lents.
Une approche microfluidique
Le laboratoire de Bart Deplancke, à l'Institut interfacultaire de Bioingénierie de l'EPFL, vient d'inventer une nouvelle technique appelée SMiLE-seq, qui peut fortement accélérer le processus. La technique utilise la microfluidique, permettant de contrôler de minuscules quantités de liquide dans des espaces aussi minuscules. La microfluidique devient un domaine d'excellence à l'EPFL, réunissant un grand nombre de domaines et de disciplines différents.
SmiLE-seq attache de petites quantités du facteur de transcription dans un microcircuit pourvu de canaux de la taille du micromètre. Une fois qu’ils sont fixés à la surface du dispositif, une grande bibliothèque d'ADN aléatoire est délicatement pompée dans celui-ci et coule sur les facteurs de transcription. Cela permet aux facteurs de transcription de reconnaître leurs séquences ADN correspondantes. Après cela, le complexe facteur de transcription-ADN est capturé en faisant descendre une touche actionnée par microfluidique, tandis que l'ADN qui n'est pas lié est lessivé.
Ensuite, l'ADN lié est retiré du dispositif et préparé en vue de son séquençage, afin d'identifier quelle partie a été capturée par les facteurs de transcription. Cette information est soumise à un logiciel spécialisé qui permet aux chercheurs de travailler sur les propriétés de liaison d'ADN des facteurs de transcription ou d'hétérodimères. En retour, cela aide à mieux prédire leurs profils de liaison d'ADN in vivo.
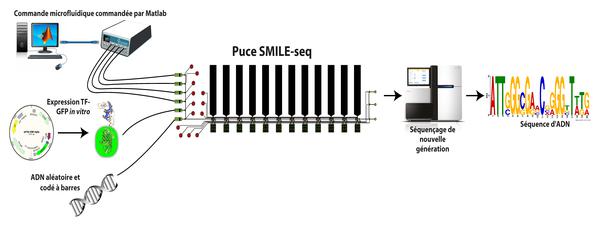
SmiLE-seq offre trois avantages principaux: d'abord, elle réduit la quantité de facteurs de transcription requis pour ce type d'expérience, puisqu'elle n'a besoin que de picogrammes de facteurs. Deuxièmement, elle accélère considérablement le processus, de plusieurs jours à moins d'une heure. Finalement, SmiLE-seq n'est limité ni par la longueur de la séquence ADN-cible, ni n'est biaisée par des interactions plus fortes d'affinités protéines-ADN.
L'équipe de Deplancke a utilisé SmiLE-seq sur 67 facteurs de transcription complets d'humains, de souris et de Drosophila, et en a analysé avec succès plusieurs qui n'avaient jamais été étudiés auparavant. A l'avenir, il entend exploiter la versatilité de cette technique dans d'autres molécules. Son équipe a déposé une demande de brevet, et une start-up est créée pour promouvoir commercialement le concept de SmiLE-seq.
Ce travail résulte d'une collaboration de l'Institut de Bioingénierie de l'EPFL avec l'Institut de Recherches expérimentales sur le cancer (EPFL-ISREC), le Globat Health institute (EPFL-GHI) et l'Institut suisse de bioinformatique. Il a été financé par le Fonds National Suisse, SystemsX.ch, et l'EPFL.
Référence
Alina Isakova, Romain Groux, Michael Imbeault, Pernille Rainer, Daniel Alpern, Riccardo Dainese, Giovanna Ambrosini, Didier Trono, Philipp Bucher, Bart Deplancke. SMiLE -seq identifies binding motifs of single and dimeric transcription factors.Nature Methods 16 January 2017. DOI: 10.1038/nmeth.4143